生物研究的一些常用在线生信工具建议收藏
生物研究的一些常用在线生信工具建议收藏
#rd
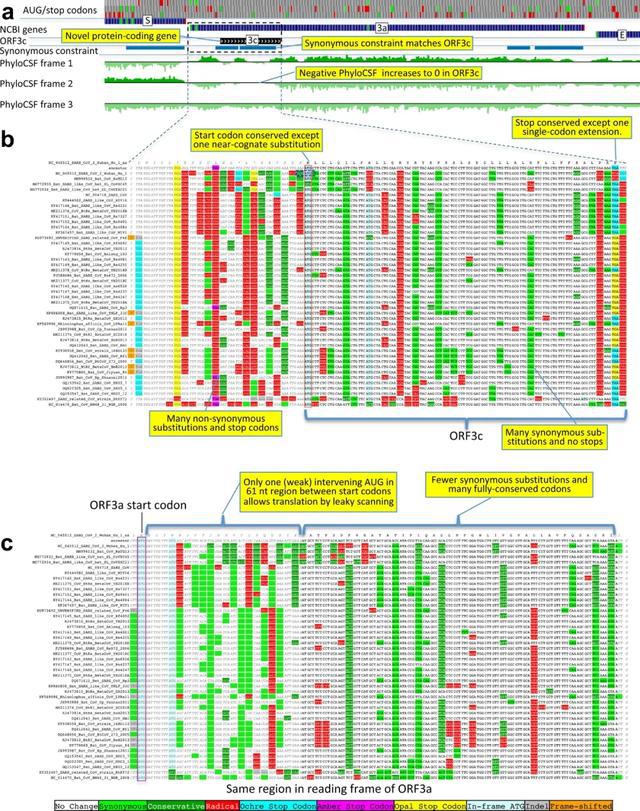
蛋白的三级结构模型预测,网站功能有蛋白序列就可以,其网址:,可以根据需要分析即可
此数据库功能比较强大,图片可以作为基本的分析用,建议收藏。
蛋白突变分析

主要是蛋白功能位点发生点突变之后,分析其稳定性的变化程度来判断是否是有害突变之类的分析。
基因组数据库
植物的基因组数据库一般用 ,其网址:,还有动物的这个网站都有,可以根据需要下载即可
此数据库格式比较规范,下载基本可以直接用,一般不建议去NCBI下载,格式一般用不直接分析,建议收藏。
在线分析网站
根据染色体起始终止点坐标来获取碱基序列 http://www.bio-info-trainee.com/1049.html
热图 http://sebastianraschka.com/Articles/heatmaps_in_r.html
热图 http://www.chibi.ubc.ca/matrix2png/
韦恩图 http://bioinfogp.cnb.csic.es/tools/venny/index.html
基因预测 http://linux1.softberry.com/berry.phtml?topic=fgenesh&group=programs&subgroup=gfind
phylogenetic http://itol.embl.de/index.shtml
启动子区预测 http://www.cbs.dtu.dk/services/SignalP/
蛋白质一级结构分析 https://www.predictprotein.org/home
蛋白质磷酸化位点 http://www.cbs.dtu.dk/services/NetPhos/
信号肽 http://www.cbs.dtu.dk/services/SignalP/
跨膜结构域 http://www.cbs.dtu.dk/services/TMHMM/
蛋白质亚细胞定位 http://psort.hgc.jp/form2.html
蛋白质二级结构分析 https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html
蛋白质三级结构预测 http://swissmodel.expasy.org/interactive
短序列拼接 http://doua.prabi.fr/software/cap3
多序列比对相似性展示 http://cotton.hzau.edu.cn/EN/tools/BioERCP/simitrix.php
绘制GO注释结果 http://wego.genomics.org.cn/cgi-bin/wego/index.pl
蛋白质domain http://pfam.xfam.org/
基因组杂合性评估 http://qb.cshl.edu/genomescope/analysis.php?code=example2
circos图 http://mkweb.bcgsc.ca/tableviewer/visualize/
序列比对muscle http://www.ebi.ac.uk/Tools/msa/muscle/#
富集分析 http://bioinfo.vanderbilt.edu/webgestalt/login.php
富集分析 http://amp.pharm.mssm.edu/Enrichr/
DNA序列过滤器 http://www.bio-soft.net/sms/filter_dna.html
序列比对 http://www.ebi.ac.uk/Tools/msa/muscle/#
在线DNA和蛋白序列处理工具集 http://www.biotrainee.com/thread-363-1-1.html
annovar对snp位点注释 http://wannovar.wglab.org/
Mutation-Assessor对突变位点对基因或者蛋白功能的影响 http://www.bio-info-trainee.com/1270.html
MutationMapper来查看基因突变的分布 http://www.bio-info-trainee.com/1270.html
GO分析 https://david.ncifcrf.gov/tools.jsp
构建进化树 http://www.atgc-montpellier.fr/phyml/
格式转换器 http://sequenceconversion.bugaco.com/converter/biology/sequences/fasta_to_phylip.php
GenBank文件特征序列提取器 http://www.bio-soft.net/sms/feat_extract.html
GenBank to FASTA http://www.bio-soft.net/sms/gen_fasta.html
随机DNA序列 http://www.bio-soft.net/sms/rand_dna.html
反序与补序工具 http://www.bio-soft.net/sms/rev_comp.html
打乱DNA序列工具 http://www.bio-soft.net/sms/shuffle_dna.html
DNA序列分组工具 http://www.bio-soft.net/sms/group_dna.html
引物显示工具 http://www.bio-soft.net/sms/primer_show.html
限制酶位点与翻译工具 http://www.bio-soft.net/sms/rest_trans_map.html
序列翻译工具 http://www.bio-soft.net/sms/show_trans.html
密码子图表 http://www.bio-soft.net/sms/codon_plot.html
密码子使用工具 http://www.bio-soft.net/sms/codon_usage.html
CpG岛 http://www.bio-soft.net/sms/cpg_island.html
DNA特征序列查找 http://www.bio-soft.net/sms/dna_pattern.html
DNA统计 http://www.bio-soft.net/sms/dna_stats.html
ORF查找器 http://www.bio-soft.net/sms/orf_find.html
位置碱基频率 http://www.bio-soft.net/sms/uneven.html
限制位点概要 http://www.bio-soft.net/sms/rest_sum.html
碱基比例图 http://www.bio-soft.net/sms/simple_plot.html
测试编码 http://www.bio-soft.net/sms/testcode.html
DNA翻译 http://www.bio-soft.net/sms/translate.html
蛋白过滤器 http://www.bio-soft.net/sms/filter_prot.html
GenBank翻译提取器 http://www.bio-soft.net/sms/trans_extract.html
随机蛋白序列 http://www.bio-soft.net/sms/rand_prot.html
一到三:单字母氨基酸残基编码到三字母编码序列 http://www.bio-soft.net/sms/oneToThree.html
打乱蛋白序列 http://www.bio-soft.net/sms/shuffle_prot.html
三到一:三字母编码的蛋白序列转换为单字母编码氨基酸的蛋白序列 http://www.bio-soft.net/sms/threeToOne.html
COMBOSA http://www.bio-soft.net/sms/combosa.html
蛋白序列分组 http://www.bio-soft.net/sms/group_prot.html
多序列对比显示 http://www.bio-soft.net/sms/multi_align.html
一致和类似性值 http://www.bio-soft.net/sms/ident_sim.html
蛋白序列统计 http://www.bio-soft.net/sms/protein_stats.html
蛋白特征序列查找 http://www.bio-soft.net/sms/prot_pattern.html
蛋白分子量 http://www.bio-soft.net/sms/prot_mw.html
Multi Rev Trans http://www.bio-soft.net/sms/multi_rev_trans.html
Reverse Translate http://www.bio-soft.net/sms/rev_trans.html
Acknowledgements http://www.bio-soft.net/sms/ack.html
Browser Compatibility http://www.bio-soft.net/sms/browser_comp.html
Download http://www.bio-soft.net/sms/d_load.html
序列比对 http://www.ebi.ac.uk/Tools/msa/muscle/#
在线DNA和蛋白序列处理工具集 http://www.biotrainee.com/thread-363-1-1.html
annovar对snp位点注释 http://wannovar.wglab.org/
Mutation-Assessor对突变位点对基因或者蛋白功能的影响 http://www.bio-info-trainee.com/1270.html
MutationMapper来查看基因突变的分布 http://www.bio-info-trainee.com/1270.html
GO分析 https://david.ncifcrf.gov/tools.jsp
构建进化树 http://www.atgc-montpellier.fr/phyml/
格式转换器 http://sequenceconversion.bugaco.com/converter/biology/sequences/fasta_to_phylip.php
网页版韦恩图:
http://bioinformatics.psb.ugent.be/cgi-bin/liste/Venn/calculate_venn.htplmatch
http://www.pangloss.com/seidel/Protocols/venn.cgi
http://bioinfogp.cnb.csic.es/tools/venny/index.html
用R中的limma包:http://www.ats.ucla.edu/stat/r/faq/venn.htm
使用方法详见:http://www.biotrainee.com/thread-105-1-1.html
可视化工具MutationMapper看基因上面突变的分布
http://www.cbioportal.org/mutation_mapper.jsp
使用方法详见:http://www.biotrainee.com/thread-388-1-1.html
用ChIP-Enrich对CHIP-seq的peaks做富集分析
paper:http://nar.oxfordjournals.org/content/early/2014/05/30/nar.gku463.long
网址:http://chip-enrich.mematchd.umich.edu/
使用方法详见:http://www.biotrainee.com/thrematchad-405-1-1.html
在线序列格式转换器(Sequence conversion)
http://sequenceconversion.bugaco.com/converter/biology/sequences/fasta_to_phylip.php
使用方法详见:http://www.biotrainee.com/thread-405-1-1.html
在线构建进化树(PhyML )
http://www.atgc-montpellier.fr/phyml/
使用方法详见:http://www.biotrainee.com/thread-399-1-1.html
GO分析
DAVID在线软件输入ENTREZ_GENE_ID(网页说明各种支持的格式)即可。https://david.ncifcrf.gov/tools.jsp
Cytoscape中安装扩展插件BINGO,不仅可以做GO分析,还可以绘制网络图。
使用方法详见:http://www.biotrainee.com/thread-401-1-1.html
Mutation-Assessor看突变位点对基因或蛋白功能影响
http://mutationassessor.org/
使用方法详见:http://www.biotrainee.com/thread-387-1-1.html
使用在线annovar对snp位点进行注释
http://wannovar.wglab.org/
使用方法详见:http://www.biotrainee.com/thread-386-1-1.html
在线DNA和蛋白序列处理工具集
详情见:http://www.biotrainee.com/thread-363-1-1.html
基因富集分析
http://amp.pharm.mssm.edu/Enrichr/
http://bioinfo.vanderbilt.edu/webgestalt/login.php
http://www.genome.jp/kegg/tool/map_pathway2.html
使用详情见:http://www.biotrainee.com/thread-90-1-1.html
网页版的热图
http://www.chibi.ubc.ca/matrix2png/
R语言:http://sebastianraschka.com/Articles/heatmaps_in_r.html
通过以上步骤是否学会了呢?关注我们,学习更多生信干货,原创不易,请各位多多点赞加转发朋友圈进行分享!
今天就先给大家介绍到这里,希望大家的科研能有所帮助!祝您科研顺利快乐!
生信漫谈
生信漫谈,认识生信,学习生信,跨越生信入门路上的障碍,从而利用生信技术解决科研学习路上的绊脚石!