机器学习特征选择—使用遗传算法进行特征选择
目录
0、前言
1、遗传算法概念
2、基于DEAP库的遗传算法特征选择
3、我的遗传算法特征选择代码及一些代码函数解析
4、完整代码
5、可能会遇到的错误
0、前言
差不多有大半年没有写博客了,这段时间没有学习什么新的知识和总结;这篇博客内容也是大概几个月前的学习内容了。趁现在有空简单总结一下,做点笔记。
因为之前我一直在学习机器学习图像处理这方面相关的知识,在我通过图像处理提取图像的信息后,接着通过这些图像信息建立机器学习回归模型,对某一个事物进行预测;在这个过程中,我偶然的一次交流会上听到了进化算法,感觉可以应用到我的研究上,从而提高模型的性能,同时,也可以观察哪些特征信息对模型的结果影响较大。
1、遗传算法概念
遗传算法提供了一种求解复杂系统问题的通用框架,它不依赖于问题的具体领域,对问题的种类有很强的鲁棒性,所以广泛应用于组合优化、机器学习、特征选择、控制科学等领域。基于遗传算法的特征选择方法由于具有良好的全局搜索能力,故而不会陷入局部最优解,同时该算法具有潜在的并行性和良好的扩展性,极易与其他方法相结合,因此基于遗传算法的特征选择方法被广泛研究。
特征选择的任务是从给定特征子集中搜索出符合条件的子特征集合,这一过程与数学中的组合优化问题类似,而遗传算法解决组合优化问题相对较好。遗传算法的搜索从种群出发,不依赖问题的具体领域,具有潜在的并行性,同时较易跳出局部极值,效率较高且性能更好。 基于遗传算法的特征选择算法具有以下优点:
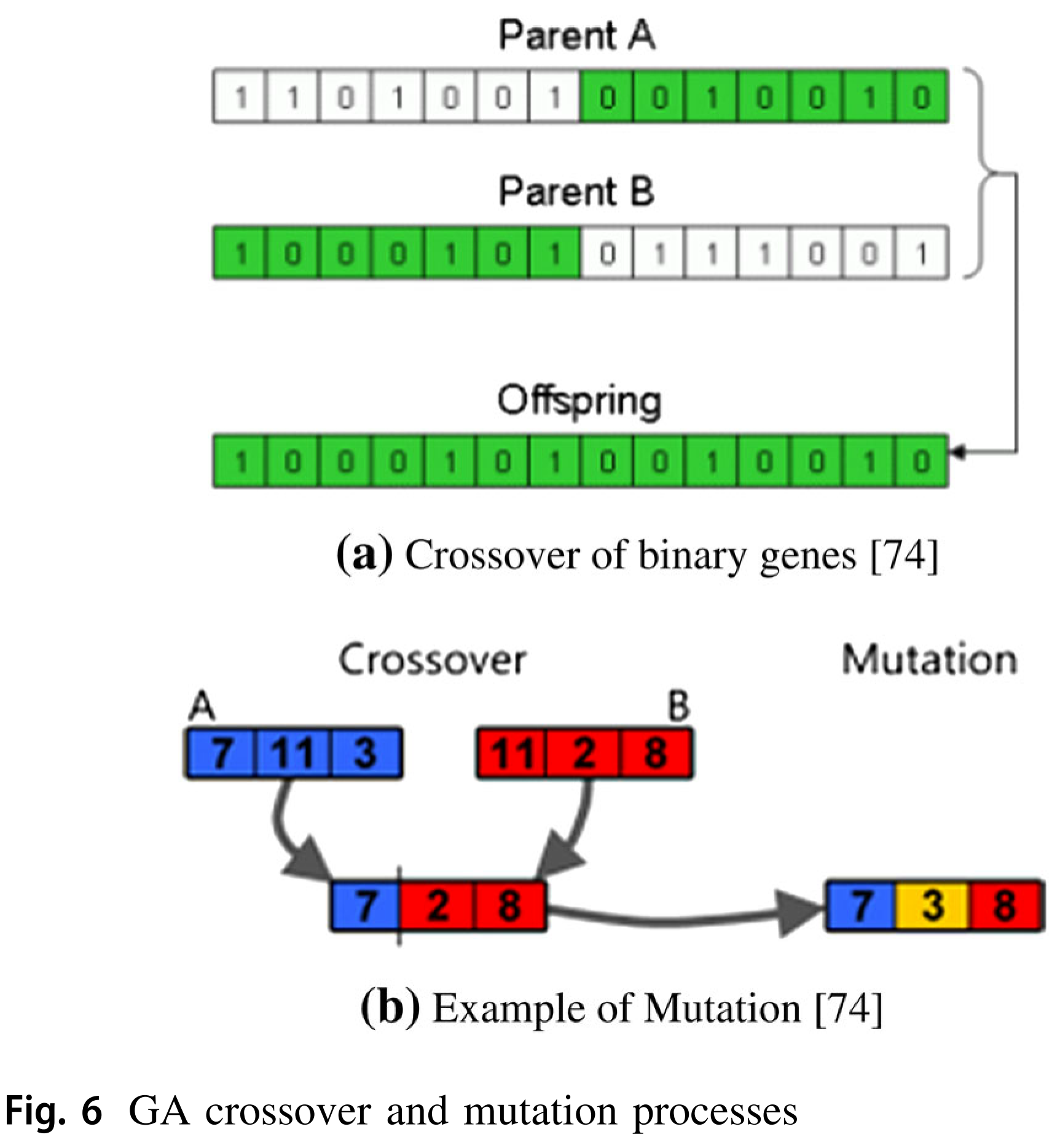
(1)遗传算法的作用对象是编码后的染色体编码串,因此与特征中数据的类型无关。对于从 M 个特征空间中选取 m 个特征的问题,可是使用由“0”和“1”组成的长度为 M 的字符串表示,“0”表示对应位的特征未被选中,“1”则表示对应特征被选中,显然字符串中“1”的个数应为 m。因此基于遗传算法的特征选择方法与目标问题的领域无关,具有较广的适用性。
(2)遗传算法的搜索过程使用适应度函数进行启发,过程简单且具有较好的可扩展性,容易与其他特征选择算法结合。
(3)相对于其他特征选择算法,基于遗传算法的特征选择方法具有良好的全局搜索能力,可以快速将解空间满足要求的个体全部搜索出来,该方法所选择出来的是一组特征,而非一个个特征的堆砌。同时该方法可以通过变异操作跳出局部最优解,因此能够有较大的概率产生全局最优解[1]。
主要的流程如下图所示:
2、基于DEAP库的遗传算法特征选择
这里的代码主要是依靠这个DEAP库实现的,具体库中的一些api和概念可以参考下面两篇博客;
进化计算-遗传算法之史上最全选择策略_松间沙路hba的博客-CSDN博客_遗传算法选择策略
基于DEAP库的进化算法-2.进化算法各元素的DEAP实现_哎呦-_-不错的博客-CSDN博客中deap
3、我的遗传算法特征选择代码及一些代码函数解析
(1)读取数据:这里是从csv文件中读取数据;
#1获取数据
#读取的特征数据,根据自己的特征数据路径和特征个数修改
data = pd.read_csv('F:/feature_data_label.csv', usecols=range(40)) # usecols=range(40)读取0-41列
# data_ = np.array(data)
#特征对应的标签,根据自己的标签路径修改
label = pd.read_csv('F:/label.csv', usecols=[1])
# label_ = np.array(label)(2)遗传算法的预估器,这里使用了一个k折验证,然对训练集和测试集进行划分和对特征编码;
# 预估器
def getFitness(individual):for train_index, test_index in kf.split(data, label):# print("Train:", train_index, "Validation:",test_index)x_train, x_test = data.iloc[train_index], data.iloc[test_index]y_train, y_test = label.iloc[train_index], label.iloc[test_index]# Parse our feature columns that we don't use解析我们不使用的特性列# Apply one hot encoding to the features 对特性应用一种热编码cols = [index for index in range(len(individual)) if individual[index] == 0]X_trainParsed = x_train.drop(x_train.columns[cols], axis=1)X_trainOhFeatures = pd.get_dummies(X_trainParsed)X_testParsed = x_test.drop(x_test.columns[cols], axis=1)X_testOhFeatures = pd.get_dummies(X_testParsed)(3)确定遗传算法的预估器,我这里使用的是MLP回归,也可以换成机器学习的其他算法;
#设置预估器并使用R2或mean_squared_error作为评判标准,这里使用是的R2
# clf = RandomForestRegressor(n_estimators=300, criterion='squared_error', n_jobs=-1, random_state=5)
clf = MLPRegressor(hidden_layer_sizes=(200, 100), activation='relu', solver='sgd', alpha=0.01, max_iter=3000,random_state=5)
# clf = svm.SVR(kernel='linear', max_iter=- 1)
clf.fit(X_trainOhFeatures, y_train.values.ravel())
y_pred = clf.predict(X_testOhFeatures)
R_2 = metrics.r2_score(y_test, y_pred)
mean_squared_error = metrics.mean_squared_error(y_test, y_pred)
scores_r2.append(R_2)
scores_mean_squared_error.append(mean_squared_error)(4)这里是DEAP库中的一些遗传算法配置,可以根据自己的需求进行更改;可以参考这篇博客,它里面有详细讲解;
# ========DEAP GLOBAL VARIABLES (viewable by SCOOP)========DEAP全局变量(可以通过SCOOP查看)
# Create Individual 创建个人
creator.create("FitnessMax", base.Fitness, weights=(1.0,))
creator.create("Individual", list, fitness=creator.FitnessMax)# Create Toolbox创建工具箱
toolbox = base.Toolbox()
toolbox.register("attr_bool", random.randint, 0, 1)
toolbox.register("individual", tools.initRepeat, creator.Individual, toolbox.attr_bool, len(data.columns))
toolbox.register("population", tools.initRepeat, list, toolbox.individual)# Continue filling toolbox...继续填工具箱# 注册遗传算法的操作:选择、交叉、突变
toolbox.register("evaluate", getFitness)
toolbox.register("mate", tools.cxOnePoint)#交叉操作
toolbox.register("mutate", tools.mutFlipBit, indpb=0.05)#突变操作,翻转突变,每个属性翻转的独立概率0.05。
toolbox.register("select", tools.selTournament, tournsize=3)#育种选择(5)这里是设置种群的大小和迭代次数;
#初始化变量
def getHof():# Initialize variables to use eaSimple使用simple初始化变量numPop = 200numGen = 80pop = toolbox.population(n=numPop)hof = tools.HallOfFame(numPop * numGen)stats = tools.Statistics(lambda ind: ind.fitness.values)(6)对选择的特征子集结果进行排序,并选择效果最好的特征子集;
# 获取对验证数据执行得最好的子集列表
maxValAccSubsetIndicies = [index for index in range(len(validation_R2_List)) if validation_R2_List[index] == max(validation_R2_List)]
maxValIndividuals = [individualList[index] for index in maxValAccSubsetIndicies]
#输出对应特征子集的标签名
maxValSubsets = [[list(data)[index] for index in range(len(individual)) if individual[index] == 1] for individual in maxValIndividuals](7)绘制曲线图;
#绘制曲线图
e = plt.figure(1)
plt.plot(percentileList, validation_R2_List, marker='o', color='r')
plt.plot(percentileList, ynew, color='b')
plt.title('Validation Set Regression R2 vs. \n Continuum with Cubic-Spline Interpolation')
plt.xlabel('Population Ordered By Increasing Test Set R2')
plt.ylabel('Validation Set R2')
e.show()f = plt.figure(2)
number = [i + 1 for i in range(len(individualList))]
plt.scatter(number, validation_R2_List)
plt.title('Validation Set Regression R2 vs. Continuum')
plt.xlabel('Population Ordered By Increasing Test Set R2')
plt.ylabel('Validation Set R2')
f.show()4、完整代码
# ****************************************************************
import pandas as pd
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import accuracy_score
from deap import creator, base, tools, algorithms
from scoop import futures
import random
import numpy
from scipy import interpolate
import matplotlib.pyplot as plt
from sklearn import svm
from sklearn import metrics
from sklearn.neural_network import MLPRegressor
from sklearn.ensemble import RandomForestRegressor
import numpy as np
from sklearn.preprocessing import StandardScaler
from sklearn.model_selection import KFold# 1获取数据
#读取的特征,需要修改相应的地址和特征个数
data = pd.read_csv('F:/feature_data_label.csv', usecols=range(40)) # usecols=range(40)读取0-41列
# data_ = np.array(data)
#特征对应的标签,需要修改相应的地址
label = pd.read_csv('F:/label.csv', usecols=[1])
# label_ = np.array(label)scores_r2 = [] #交叉验证r2的得分
scores_mean_squared_error = [] #交叉验证均方误差的得分
Select_time = [] #遗传算法选择某个特征的总次数
#K折交叉验证
kf = KFold(n_splits=5, shuffle=True,random_state=5) # 5折交叉验证
# 2数据集划分
# x_train, x_test, y_train, y_test = train_test_split(data, label, test_size=0.2, random_state=5)# 预估器
def getFitness(individual):for train_index, test_index in kf.split(data, label):# print("Train:", train_index, "Validation:",test_index)x_train, x_test = data.iloc[train_index], data.iloc[test_index]y_train, y_test = label.iloc[train_index], label.iloc[test_index]# Parse our feature columns that we don't use解析我们不使用的特性列# Apply one hot encoding to the features 对特性应用一种热编码cols = [index for index in range(len(individual)) if individual[index] == 0]X_trainParsed = x_train.drop(x_train.columns[cols], axis=1)X_trainOhFeatures = pd.get_dummies(X_trainParsed)X_testParsed = x_test.drop(x_test.columns[cols], axis=1)X_testOhFeatures = pd.get_dummies(X_testParsed)# 3特征工程标准化transfer = StandardScaler()X_trainOhFeatures = transfer.fit_transform(X_trainOhFeatures)X_testOhFeatures = transfer.transform(X_testOhFeatures)#设置预估器并使用R2或mean_squared_error作为评判标准,这里使用是的R2# clf = RandomForestRegressor(n_estimators=300, criterion='squared_error', n_jobs=-1, random_state=5)clf = MLPRegressor(hidden_layer_sizes=(200, 100), activation='relu', solver='sgd', alpha=0.01, max_iter=3000,random_state=5)# clf = svm.SVR(kernel='linear', max_iter=- 1)# clf = MLPRegressor(hidden_layer_sizes=(200, 100), activation='relu', solver='sgd', alpha=0.01, max_iter=12000,# random_state=5)clf.fit(X_trainOhFeatures, y_train.values.ravel())y_pred = clf.predict(X_testOhFeatures)R_2 = metrics.r2_score(y_test, y_pred)mean_squared_error = metrics.mean_squared_error(y_test, y_pred)scores_r2.append(R_2)scores_mean_squared_error.append(mean_squared_error)scores_r2_mean = np.mean(scores_r2)scores_mean_squared_error_mean = np.mean(scores_mean_squared_error)scores_r2.clear()scores_mean_squared_error.clear()return (scores_r2_mean,scores_mean_squared_error_mean)# ========DEAP GLOBAL VARIABLES (viewable by SCOOP)========DEAP全局变量(可以通过SCOOP查看)
# Create Individual 创建个人
creator.create("FitnessMax", base.Fitness, weights=(1.0,))
creator.create("Individual", list, fitness=creator.FitnessMax)# Create Toolbox创建工具箱
toolbox = base.Toolbox()
toolbox.register("attr_bool", random.randint, 0, 1)
toolbox.register("individual", tools.initRepeat, creator.Individual, toolbox.attr_bool, len(data.columns))
toolbox.register("population", tools.initRepeat, list, toolbox.individual)# Continue filling toolbox...继续填工具箱# 注册遗传算法的操作:选择、交叉、突变
toolbox.register("evaluate", getFitness)
toolbox.register("mate", tools.cxOnePoint)#交叉操作
toolbox.register("mutate", tools.mutFlipBit, indpb=0.05)#突变操作,翻转突变,每个属性翻转的独立概率0.05。
toolbox.register("select", tools.selTournament, tournsize=3)#育种选择
# ========
#初始化变量
def getHof():# Initialize variables to use eaSimple使用simple初始化变量numPop = 200numGen = 80pop = toolbox.population(n=numPop)hof = tools.HallOfFame(numPop * numGen)stats = tools.Statistics(lambda ind: ind.fitness.values)stats.register("avg", numpy.mean)stats.register("std", numpy.std)stats.register("min", numpy.min)stats.register("max", numpy.max)# Launch genetic algorithm启动遗传算法pop, log = algorithms.eaSimple(pop, toolbox, cxpb=0.5, mutpb=0.2, ngen=numGen, stats=stats, halloffame=hof,verbose=True)# Return the hall of famereturn hofdef getMetrics(hof):# Get list of percentiles in the hall of famepercentileList = [i / (len(hof) - 1) for i in range(len(hof))]# percentileList = [i for i in range(len(hof))]
#对选择的特征子集进行排序# Gather fitness data from each percentile# test_R2_List = []validation_R2_List = []individualList = []for individual in hof:# test_R2 = individual.fitness.valuesvalidation_R2 = getFitness(individual)# test_R2_List.append(test_R2[0])validation_R2_List.append(validation_R2[0])individualList.append(individual)# test_R2_List.reverse()validation_R2_List.reverse()individualList.reverse()return validation_R2_List, individualList, percentileList# return test_R2_List, validation_R2_List, individualList, percentileListif __name__ == '__main__':'''First, we will apply logistic regression using all the features to acquire a baseline accuracy.'''individual = [1 for i in range(len(data.columns))]Test_R2 ,Test_MSE = getFitness(individual)# validationAccuracy = getFitness(individual, X_trainAndTest, X_validation, y_trainAndTest, y_validation)print('\nTest R2 with all features: \t' + str(Test_R2))print('Test MSE with all features: \t' + str(Test_MSE))# 现在,我们将应用遗传算法来选择一个特征子集,它给出了比基线更好的准确性hof = getHof()# test_R2_List, validation_R2_List, individualList, percentileList = getMetrics(hof)validation_R2_List, individualList, percentileList = getMetrics(hof)# 计算每个特征被选择的次数individualList_transpose = list(map(list, zip(*individualList))) # 转置for i in range(len(individualList_transpose)):Select_time.append(individualList_transpose[i].count(1))print(Select_time)# 获取对验证数据执行得最好的子集列表maxValAccSubsetIndicies = [index for index in range(len(validation_R2_List)) if validation_R2_List[index] == max(validation_R2_List)]maxValIndividuals = [individualList[index] for index in maxValAccSubsetIndicies]#输出对应特征子集的标签名maxValSubsets = [[list(data)[index] for index in range(len(individual)) if individual[index] == 1] for individual in maxValIndividuals]print('\n---Optimal Feature Subset(s)---\n')for index in range(len(maxValAccSubsetIndicies)):# print('Percentile: \t\t\t' + str(percentileList[maxValAccSubsetIndicies[index]]))print('Validation R2: \t\t' + str(validation_R2_List[maxValAccSubsetIndicies[index]]))print('Individual: \t' + str(maxValIndividuals[index]))print('Number Features In Subset: \t' + str(len(maxValSubsets[index])))print('Feature Subset: ' + str(maxValSubsets[index]))# 现在,我们绘制测试和验证分类准确性的图表,看看当我们从最差的特征子集移动到利用遗传算法找到最佳特征子集。# 计算验证分类精度的最佳拟合线(非线性)tck = interpolate.splrep(percentileList, validation_R2_List, s=5.0)ynew = interpolate.splev(percentileList, tck)#绘制曲线图e = plt.figure(1)plt.plot(percentileList, validation_R2_List, marker='o', color='r')plt.plot(percentileList, ynew, color='b')plt.title('Validation Set Regression R2 vs. \n Continuum with Cubic-Spline Interpolation')plt.xlabel('Population Ordered By Increasing Test Set R2')plt.ylabel('Validation Set R2')e.show()f = plt.figure(2)number = [i + 1 for i in range(len(individualList))]plt.scatter(number, validation_R2_List)plt.title('Validation Set Regression R2 vs. Continuum')plt.xlabel('Population Ordered By Increasing Test Set R2')plt.ylabel('Validation Set R2')f.show()
5、可能会遇到的错误
假如在运行程序时,遇到下图这样的错误时,可以按照以下步骤进行修改,具有原理我暂时也还没有了解清楚。
步骤1:点击下图报错的文件链接,进入base.py文件:
步骤2:进入base.py文件后,假如是点击上面的链接,会自动跳转到base.py文件程序的187行,然后注释掉下面红色框的这行代码(188行)就可以了。